RStudioでGitを使う
初めに
前回の記事で、RStudioでRを開発することにしました。
RStudioではGitでversion管理を行えるらしいので、Gitを入れてみます。
Gitを入れる
Gitのページからダウンロードします。
ほぼこのブログを参考にGit for Windowsを入れました。
ブログと違うところは、
Choosing the default editor used by GitをUse Visual Studio Code as Git's default editorにしたこと
Choosing HTTPS transport backendをUse the native Windows Secure Channel libraryにしたこと
Configuring the line ending conversionsをCheckout Windows-style, commit Unix-style line endingにしたこと
くらいです。
プロンプトで行った設定↓
$ git --version git version 2.28.0.windows.1 $ git config --global user.name "wet-to-dry" $ git config --global user.email "*****@*****.com" $ git config --list diff.astextplain.textconv=astextplain filter.lfs.clean=git-lfs clean -- %f filter.lfs.smudge=git-lfs smudge -- %f filter.lfs.process=git-lfs filter-process filter.lfs.required=true http.sslbackend=schannel core.autocrlf=true core.fscache=true core.symlinks=true pull.rebase=false credential.helper=manager core.editor="C:\Users\*****\AppData\Local\Programs\Microsoft VS Code\Code.exe" --wait user.name=wet-to-dry user.email=*****@*****.com
RStudioでGitを使う
今度はこのブログを参考に進めていきます。
まず、File>New Projectから新しいプロジェクトを作成しますが、図の「Create a git repository」にチェックを入れておきます。

すると、右上にGitのタブができます。
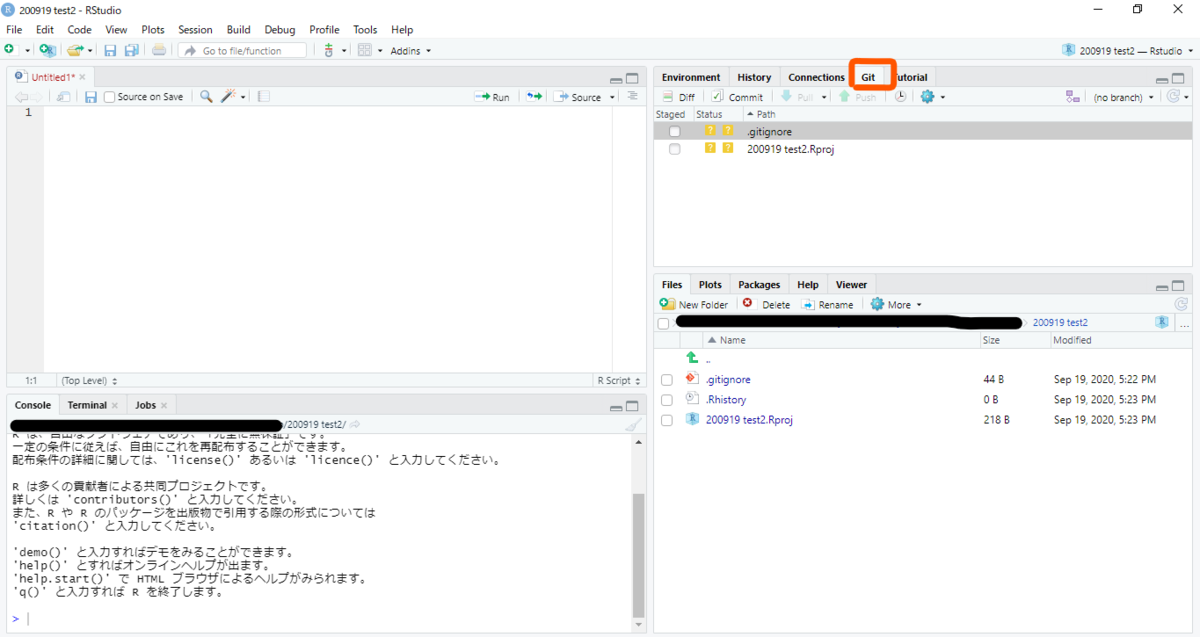
Gitペイン内のDiffボタンを押すと、commit用のウインドウが出てきます。
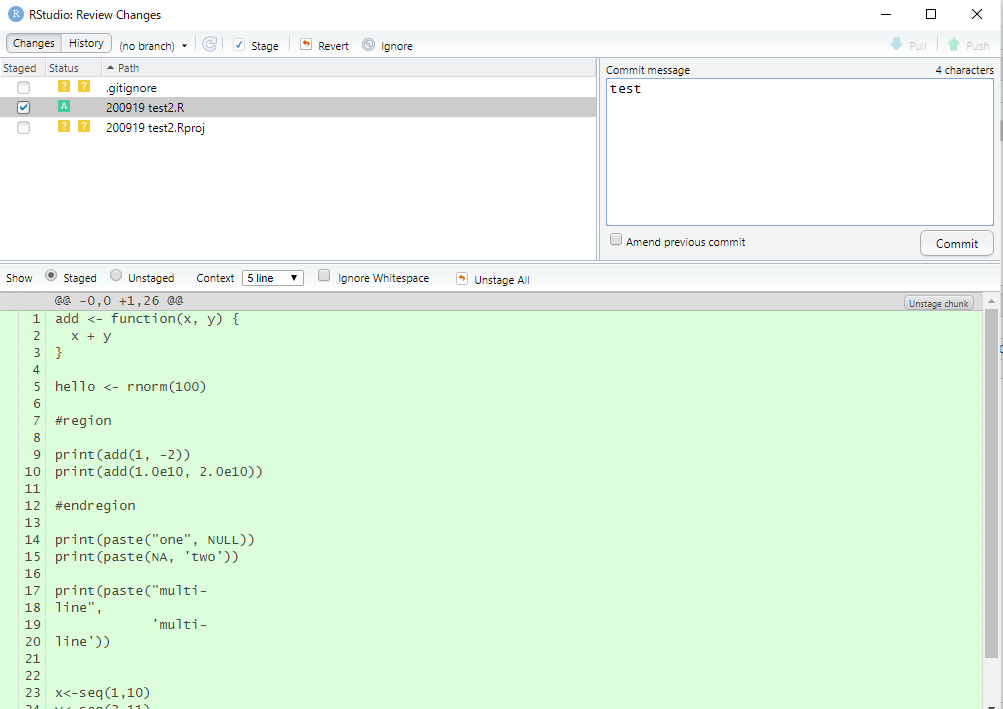
差分のcommitバージョン。

ちなみに、Status内のマークは
?→gitの管理対象になっていない
A(Added)→新たにgitの管理対象になった
D(Delete)→リポジトリにあったファイルが削除された
M(Modified)→リポジトリの状態から変更されている
U→リポジトリ間で差分が衝突している
らしいです。
また、フォルダ内に.gitignoreというファイルが作られますが、これはgitの管理対象にしないファイル(Gitペインに表示されないファイル)のリストらしいです。これを編集すれば管理対象にしないファイルを指定できるらしい。
GitHubと連携
引き続きこのブログを参考に進めていきます。
今までの処理はすべてローカルで保存されているだけらしい。
なので、GitHubなどのリモートレポジトリに入れられるようにする。
Rstudio用の新しいレポジトリを作成し、"https://github.com/ユーザー名/レポジトリ名.git"をコピー。

RstudioのGitペイン上で、歯車のボタンからShell...をクリック
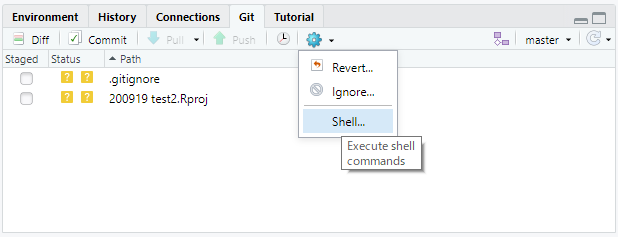
出てきたWindows Power Shellに
git remote add origin https://github.com/ユーザー名/レポジトリ名.git
を打ち込みます。
その後、branchボタンから
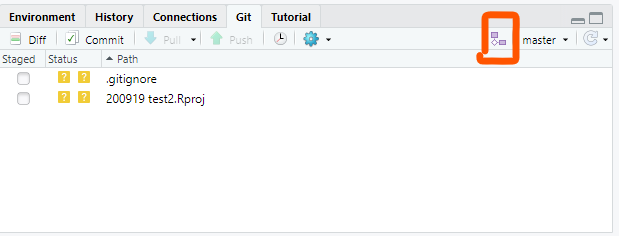
新しいbranchを作成し(Remoteはoriginでいいです)、Create!
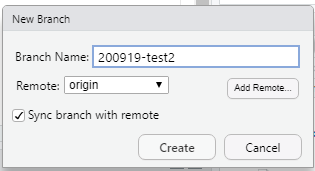
こんな感じで新しいbranchができます。
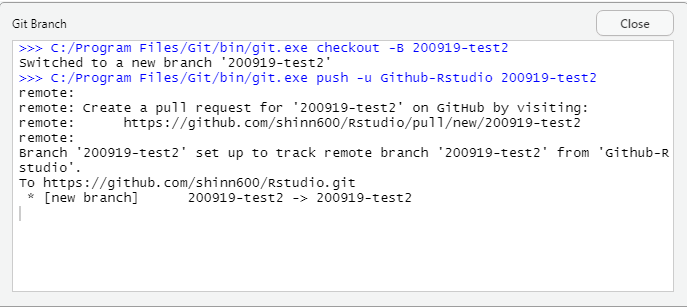
変更点がcommitし終わったら、pushボタンでリモートレポジトリを変更!
感想
とりあえずうまく行ったので一安心。
参考
研究用に使うRの開発環境(Rstudio vs. VSCode)
初めに
こんにちは。お久しぶりです。
以前Rを使ってプロット作成の練習などはしたのですが、その後特に必要性を感じずに研究生活を送ってきました。
最近統計処理をするのにt検定だけじゃ足りなくなって来て、本格的にプログラムを組んだ方がいいかなと思ったので、Rに再度手を出してみました。
型から入れとよく言いますので(プログラミングでは逆だという方もいるかもしれませんが)、まず開発環境を整えようと思います。
以前Rを触った時にはRといえばRStudioという認識だったんですが、ネットで検索するとVSCodeでできるようだったので、VSCodeとRstudioを比較してみました。
ちなみに、以前僕がVScodeをinstallした記事はこちら↓
installと事前準備
Rのinstall
2022/07/02追記 RやRstudioのインストールはProgramFileではなくUserディレクトリの方が良い。参照↓
ここからinstallしてください↓
参考ブログ (ここにも一応RStudioのinstallの仕方が書いてあります)
RStudio
以下のサイトから"RStudio Desktop"をダウンロード*1。
Rstudioを起動するとこのような画面が出てくるはず↓

まず、File>New Project>New Directory>New Projectでディレクトリを作成する場所を指定する。
指定した名前の新しいディレクトリと、その内部に新しい.Rprojファイル(ここでは"200919 test.Rproj")が作成される。

これでcurrent directoryが指定したディレクトリに変更されていればOK(図中赤色)。
次にFile>New File>R scriptを選択し、新しいscript画面を開きます。
File>Save As>で、作成したscriptも同じ場所に保存します("200919 test.R")。
これで準備は終わり。
VSCode

次に、設定から
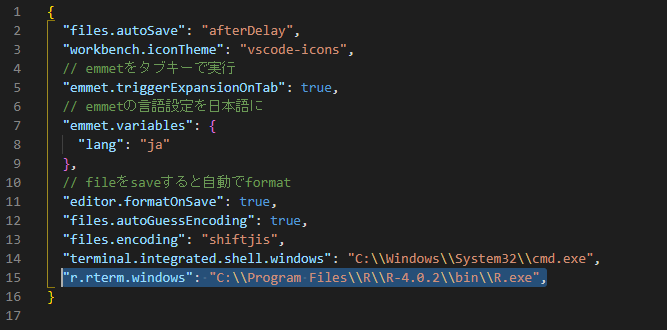
右上のJSONを開くをクリック
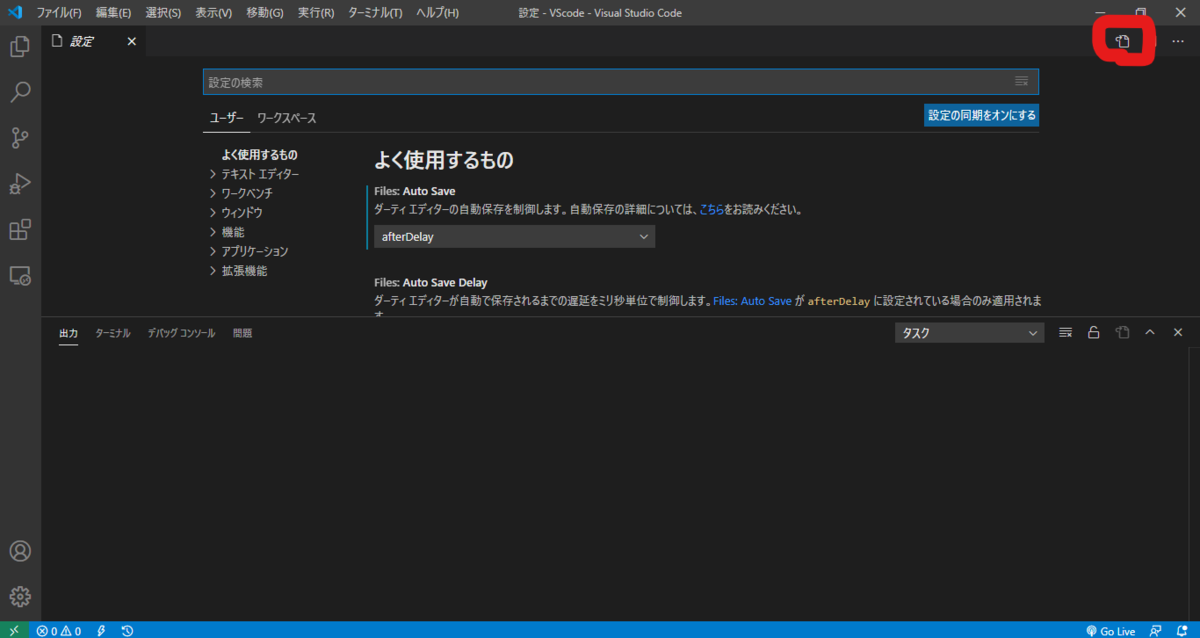
JSONの一番下に
"r.rterm.windows": "C:\\Program Files\\R\\R-4.0.2\\bin\\R.exe",
を書き加えます(PATHやRのversionは自分のRを確認して!)。
これで準備は終わり。
実際に動かしてみる
こちらのブログから、デモ用のコードを拝借して使っています。
add <- function(x, y) { x + y } hello <- rnorm(100) #region print(add(1, -2)) print(add(1.0e10, 2.0e10)) #endregion print(paste("one", NULL)) print(paste(NA, 'two')) print(paste("multi- line", 'multi- line')) x<-seq(1,10) y<-seq(2,11) plot(x, y)
RstudioもVSCodeも、
Ctrl+Enterで一行ずつ実行
Ctrl+Shift+Enterで全ての行を実行
です。うまく動けばOK!

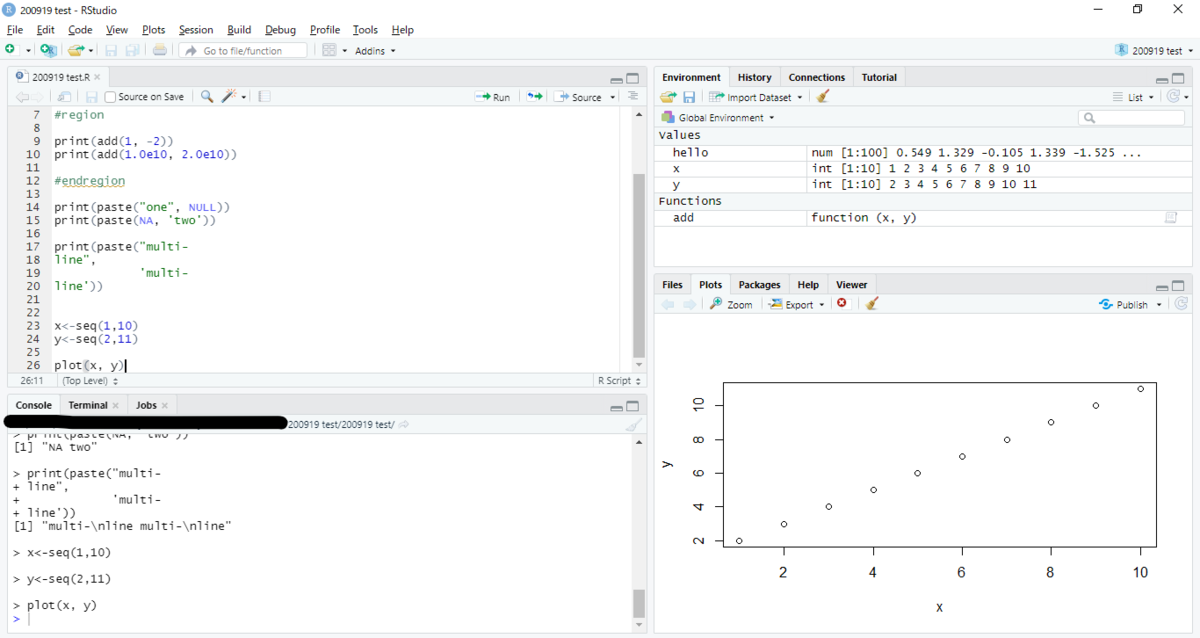
VSCodeではplotを作成する時、違うソフト(R)のダイアログとして作成します。
pythonなどの他の言語と違い、Rでの解析はビッグデータなどの数値データを扱うことが多いと思います。
また、データに合わせてコーディングしなければならないため、その場でコードを書きなおすことも多いのではないでしょうか。
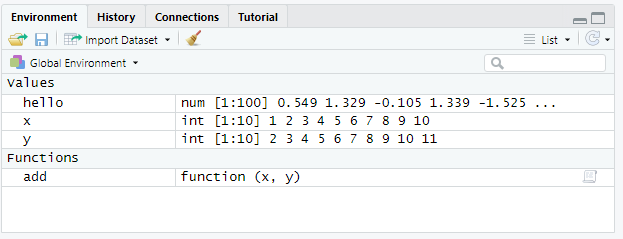
こうなると、どの変数に度の値が入っているかをすぐにチェックするために「環境変数の一覧」を見ることがあります。
RStudioでは右上にこの環境変数「Environment」ペインが存在します。
一方、VSCodeでは「F1」キーを押し、「R: Preview Environment」を開かなければなりません。
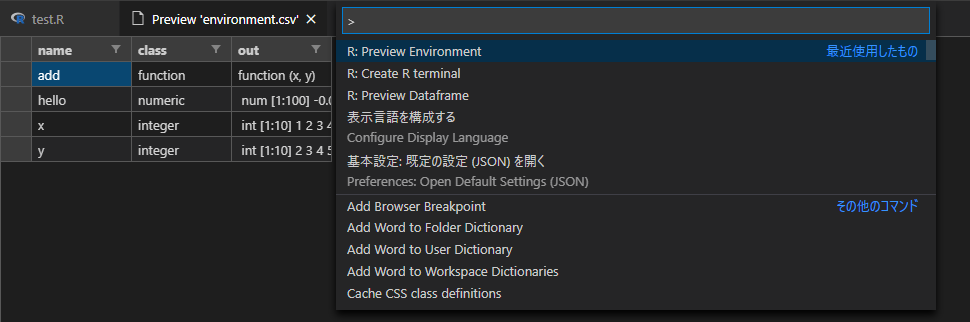
この環境変数の確認方法はrealltimeで値を更新してくれるわけではないので、毎回主導で更新しなければなりません。
比較
VSCodeは多言語と開発環境が揃えられるメリットがあるが、RStudioの方が環境変数の確認が便利という感じでした。また、スクリプトファイルの保存方法がRStudioの方が若干面倒かな、とも思いました(.Rprojファイルが多い)。
非常に甲乙つけがたかったのですが、今のところはRStudioを使っています。グラフのExportがしやすいのと、環境変数が見やすいのが大きいですね。また詰まったらVSCodeに乗り換えます。
次はGitとのリンクとかを勉強しよう。
参考
*1:"RStudio Server"はサーバー機能があるRStudioらしいのですが、僕は自分のデータを解析するだけなので通常のDesktop版でOK
OneNoteを電子実験ノートとして使う方法
Onenoteって便利なの?
現在、バイオ系の研究室で博士後期課程の学生をしております。
よく写真を撮って実験ノートに貼っていたのですが、せっかく電子データで写真を取得するのになぜ印刷して貼らねばならないのかといつも思っていました。
電子実験ノートなるものが存在することは知っていたのですが、調べてみると基本的に企業向けの有償プランが多く、無償プランであるBenchlingの電子実験ノート機能を使ってみたのですが*1*2存外不便だったので結局は紙のノートに戻ってしまいました。
数か月前、Twitterである研究者の方がOnenoteが便利だとおっしゃっていたので、Onenoteで実験ノートをとってみたところ、今まで紙でノートをとっていた自分がバカらしくなるほど便利だったので、導入過程と注意点を共有します。
OneNoteにプロトコールも実験結果も全部書いておいて、修正はすべて取り消し線と追記にしているので、『どのプロトコールがどのように変化していったか』がわかるので、どの辺がクリティカルだったかすぐにわかるんだよね pic.twitter.com/xWLAnD5DPG
— トクロンティヌス (@tokurontinus) 2020年6月19日
電子実験ノートとは
電子実験ノートと一言にいっても、いろいろ制約があります。
下のリンクの資料にも書いてありますが、「網羅性、検索性,保全性,実証性」を持っている必要があります。
以下は僕がまとめたものです。
網羅性ーーーすべての情報を不足なく記述していないと、特許出願の際に支障が出ます。また、前の実験を見返すときに、以前は重要だと思っていなかったものが実は重要だったというようなこともあるので、できる限り情報は残さないといけません。
検索性ーーー過去の実験を見返すときに、1時間も2時間もかかっていては実験が捗りませんし、見返したくなくなります。ほしい情報をすぐに手に入れることができることも重要です(サイトの検索バーみたいなものが実験ノートあれば最高ですよね!)。
保全性ーーー紙の実験ノートは1冊だけなのでデータ漏洩の観点から言えば便利ですが、逆に言うと原本を無くすと手の施しようがありません。また、貼った糊がはがれてしまったり、文字がかすれて読めなくなったりすることがあります。電子実験ノートでは、紙のものよりも長い間保存できる必要があります。
実証性ーーー実験ノートは信用に足るものでなければなりません。実験者と実験責任者が内容に同意してサインすることが決まっていますし、記載後に改ざんされた可能性があってはいけません。
人によって、ここに「操作性」などを入れることもありますが、基本的にはこれらを満たしていればOKということになります。
網羅性に関しては、自分で頑張るしかないと思います。検索性に関しては、OCRもあるのでOnenoteは最上位ではないでしょうか?保全性も、クラウド上に原本があるので、Microsoftに見られる可能性を除けば◎。実証性に関しては紙のノートに比べて少し劣るかな?という感じです。
Onenoteを電子実験ノート化する論文???
あるサイト*3にマイクロソフトOneNoteを電子実験ノートとして使う方法と題して一つの論文が紹介されていました。
こちらがその論文で、「A quick guide for using Microsoft OneNote as an electronic laboratory notebook (2019)」は電子実験ノート(ELN(Electronic Laboratory Notebook))を研究しているグループの論文です(open access)。
彼らは以前、様々な電子実験ノートを比較した経験*4を生かして、Onenoteのおすすめのカスタマイズ方法を提案しています(この論文では、驚くべきことに市販の電子実験ノート有償プランよりOnenoteの方が優れている点が多かったと述べられています)。
CC BY 4.0でしたので、僕が日本語訳したものも共有しておきます。
電子実験ノートに限らないこともありますが、以下僕の大事だと思ったことをいくつかメモします。
まず、論文内にあったまとめの表の日本語版

また、
実験やプロトコルなどの登録時には、時間の経過とともに追跡可能な重要な情報をラベル付けしておく必要がある。
例えば、実験にはEX_0001_SG_29/08/2018. HeLa細胞のRNA抽出という名前を付けることができる。
ここで、EX=実験、0001=実験番号、SG=研究者のイニシャル、29/08/2018=登録日、およびHeLa細胞のRNA抽出=実験のタイトルである。
さらに、実験、プロトコル、バイオインフォマティクス解析のいずれかを、実験=EX、プロトコル=PR、バイオインフォマティクス=BIと適宜ラベリングしてもよい。
さらに、
内部的には、同じ研究室のメンバー間でELNを共有することができ、研究室のメンバーによる不慮の変更を避けるために、「閲覧可能」モードの下でELNを共有することができる。
法的文書、ページ、またはセクション全体をパスワードで保護することで、機密情報を保護することができる(OneNote > 校閲 > パスワード)。
最後に、
ERES(電子記録と電子署名)については、OneNoteではノートに電子署名するオプションはないが、完成した実験やプロトコールなどはpdf形式でエクスポートして電子署名することができる。
これらに気を付けてやっていきます。
また、当該論文内で電子実験ノートを導入するのに役に立ちそうな引用論文がありましたので、そちらも紹介しておきます。
「Ten Simple Rules for Digital Data Storage (2016)」
https://journals.plos.org/ploscompbiol/article?id=10.1371/journal.pcbi.1005097
「Ten Simple Rules for a Computational Biologist’s Laboratory Notebook(2015)」
https://journals.plos.org/ploscompbiol/article?id=10.1371/journal.pcbi.1004385
実際に使ってみて
こんな感じで使っています。
黒塗りばっかですみません。。。
一応、まだ身バレしてないと思うので実験内容で研究室を絞り込まれないように伏せさせていただきます。
これじゃよくわからないという人は、自分でやってみて!
その他設定
その他、自分で思ったことをメモっていますので、それをコピペします。
Onenoteの構造
ノートブック→***のノートブック(1つ)
(セクショングループ→必要があれば。それぞれの研究テーマ)
セクション→実験内容、プロトコル、研究アイデア
ページ→それぞれの実験(日付別)、プロトコル(種類別)
(サブページ→実験を細分化するときに臨時で使う)セクショングループ、サブページ機能は今のところ使っていない
ページ内容
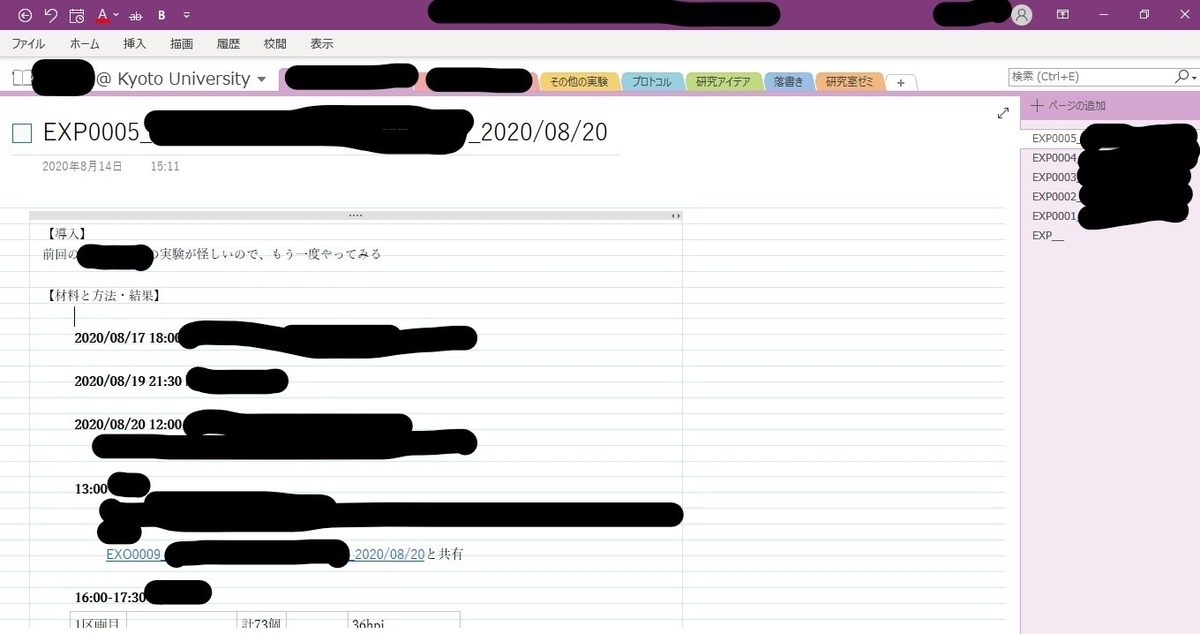
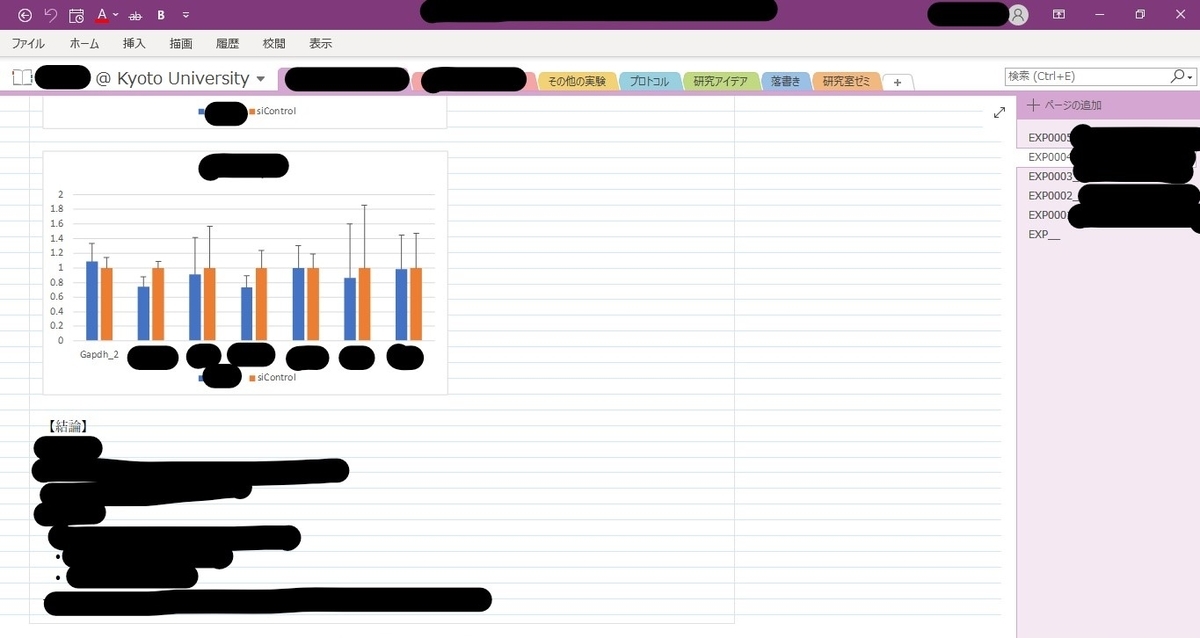
実験用のページ内容
名前:EXP0001○○○○2020/01/01
【導入】仮説、背景、目的
【材料と方法】使用するプロトコル、サンプル名、試薬名
【結果】結果、実験の変更点、気づいたこと
【結論】結論、考察、次回の展望- EXPの「P」はテーマ別に変える。僕はEXP, EXG, EXOを使ってる(それぞれ実験テーマの頭文字)
プロトコル用のページ内容
名前:PR0001××××2020/01/01
【材料】必要な試薬、必要な機材
【内容】実際の手順
【コピペ用】実験用に張り付けるための情報
電子署名
(Adobe Acrobatを持っていること前提)
※法的拘束力があるかどうかは確認を取っていません
1. ページをエクスポート(OneNote→印刷→プリンターはMicrosoft Print to PDFまたはAdobe PDFを選択、ページは「現在のページ」を選択、拡大縮小は「ページに合わせる」を選択→印刷ボタンを押す)
2. 保存フォルダを選択
3. フォルダ内に保存されるので、ファイルをAdobe Acrobatで開く
4. ツール→証明書→電子署名を押し、PDFの空いている箇所をドラッグで選択する
5. デジタルIDで署名→続行→署名をクリック
6. 保存先を選択する(僕は上書き保存している)
その他、気を付けていること
- 大容量の生データがあるときはリンクを貼る(シーケンスの.ab1、Tiff、seqデータなど)
- 先生とのディスカッションなども書き込む
- ネガティブデータもすべて書き込む
- 基本的にセクションはすべてパスワードを付けて保存(OneNote>校閲>パスワード)
- 日をまたぐ実験を一つのページに書く際は、タイムスタンプ
- 日付はAlt+Shift+Dで、時間はAlt+Shift+Tでも出る
- ページ内に複数のノートボックスを作らない
- 図や表は、キャプションや詳細な情報を添えて説明する
- 間違いはdeleteではなく、
取り消し線で消す - プロトコルは共有の利便性も鑑みて、Wordファイルで元作成→Onenoteにコピペする
バイオインフォマティクス解析
- in sillico解析では、分析に時間がかかる解析は中間データを残しておく
- in sillico解析では、使用したコードも忘れず記入する
バックアップ
- バックアップは必ず取る(Onedrive、PC、ハードドライブの3つ)
おすすめの設定
- 表示タブから罫線(最小)か方眼紙(最小)を入れるとノート感が出ておすすめ
- フォントは游ゴシック10.5ptがおすすめ
- Onenoteには有名なバグがあり、既定のフォントをいじっても文章を打っている途中で游ゴシックに変換されてしまうので、あらかじめ游ゴシックにしておくのがベター
OneNoteで設定しているフォントに関係なく游ゴシックが表示されてしまう - マイクロソフト コミュニティ
まとめ
Onenote、めちゃくちゃ快適です。
ただし、中途半端にやるとか、ボスの許可なくやるとかはしないでください。
責任は負いかねますので。
*1:Benchlingはプラスミド編集するのに便利なツールで、Apeの上位互換みたいなやつ。
*2:一応ボスに確認して許可はもらいました。
*3:http://scienceandtechnology.jp/archives/30792
*4:https://journals.plos.org/plosone/article?id=10.1371/journal.pone.0160428
生物系論文のまとめ方について~フォーマットと保存方法~
あれ?この論文どんな内容だっけ?
論文を読むときは、PCFを「論文」フォルダに入れるようにしていたのですが、ラボ配属から5年目にもなって来るとPDFがすごい数になります。 (今数えたら300本くらいあった、、、)
今まではフォルダを分けたりして何とかしてきましたが、そろそろ限界に近づいてきています。
加えて、昔読んだ論文で、
- この論文の目的なんだったっけ?
- 結論なんだっけ?
- このfigの説明でなんて言ってたか思い出せない、、、!
なんて思うことが増えてきました。
もちろん読み返せばいいんですよ?一度読んだ論文だからパパっと読み飛ばせるし、1分もかからないうちに「ああ、こういうことだった」ってなります。
今のうちはね、、、
絶対これ数年後には読んだことすら忘れてるやつやん。
ということで、論文のまとめ方を試行錯誤します。
落合先生のフォーマット
この記事を書こうと思ったのにはきっかけがあって、かの有名な筑波大学の落合陽一先生が論文のまとめ方フォーマットを公開していたんですよね(48スライド目)。
www.slideshare.net
このまとめ方はいろいろな人が評価してて、実際とてもいいと思うんですよ。
このシステム、メリットは大きく二つあって、一つは後で論文を読み直すときにすでにまとめられたものを読むので要点がつかみやすいこと。もう一つは、最初に論文を読むときにフォーマットを埋めようと意識することで、比較的早い速度で読み込むことができるということ。という風に、一粒で二度おいしいのです。
さらにこのフォーマット、意図してかはわかりませんが、論文の体裁をほとんど保ったまま要約できるのです。
どんなもの?→Abstract
先行研究と比べてどこがすごい?→Introduction
技術や手法のキモはどこ?→Material&Method
どうやって有効だと検証した?→Result
議論はある?→Discussion
次に読むべき論文は?→Reference
結構忠実に守ってますよね。
生物系用のフォーマットづくり
しかし、落合先生は機械工学の方。バリバリの生物系である僕は、このフォーマットを生物系論文用にちょこっと編集させていただきます。
1. 生物系はデータが多い
これは主にCell系列のジャーナルに言えることですが、生物系はFig6とかFig8とか普通に出てくるくらい、データ量がものすごいです。生物系は実験誤差が大きいので一つのことを言うのに二つとか三つの手法で確かめるなど、回りくどい説明が必要であることに加えて、「遺伝子カスケード」なる複数の因子が重なり合うものを証明しようとすることがあります。
これらを1ページA4にまとめようとするのは至難の業ですし、早く論文を読むという目的に対して労力が見合いません。
2. 論文内容のdiagram化
diagramとは、よく論文のFigの最初や最後にあるような、論文の概略を一枚の図(いわゆるポンチ絵)にしたものです。1.とも関連しますが、データがとても多い場合、これがあるのとないのとでは論文を読むスピードが格段に違います。もはやdiagramがあれば勝ったと言っても過言ではありません。
なければ作るのがベストです。確かに作図はめちゃくちゃめんどくさいですが、作る過程で論文の内容を自分なりに噛み砕くことができ、より深い理解にもつながります。
1 & 2 の理由から、Figはdiagramのみに一本化しようと思います。また、「これが一番大事」というチャンピオンデータがあれば、入れることができるようにフレキシブルにしておきます。
3. 生物系はマテメソがあまり重要でない
これは色々議論があるかもしれませんが、僕は生物系の論文はマテメソがとても画一的だと思っています。ウエスタンブロッティング、蛍光染色、RNA-seqなどなど、名前を聞けば大体わかってしまうようなものが多いです。Method系の論文を除き、マテメソを読み飛ばす人は多いと思いますし、実際再現実験などを行うとき以外はあまり詳細は問題になりません。
生物種と実験手法名だけを書くことも多いと思うので、他の欄とひとくくりにしてしまいましょう。
以上を加味して作成したのがこちら↓
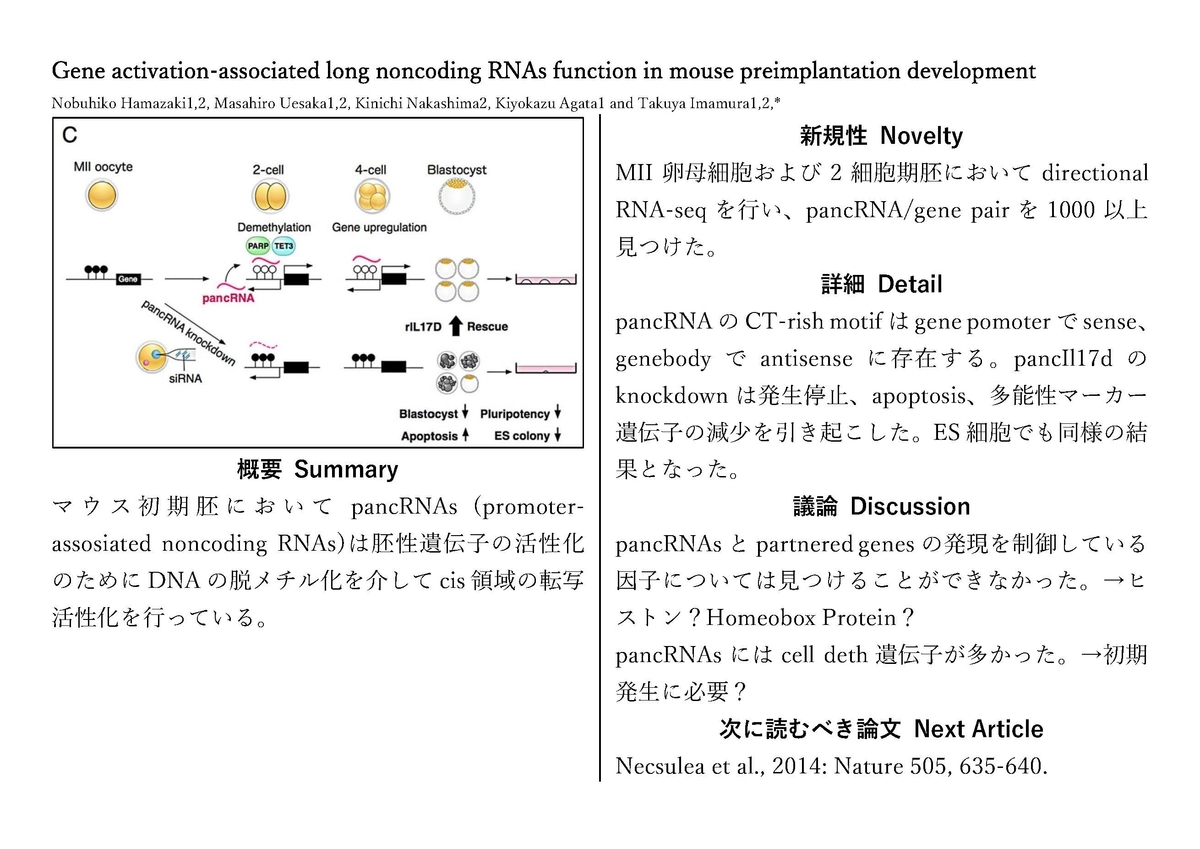
論文は、CC BY 3.0ライセンスだった
Hamazaki, N., Uesaka, M., Nakashima, K., Agata, K. and Imamura, T. (2015) Gene activation-associated long noncoding RNAs function in mouse preimplantation development. Development 142, 910-920 doi:10.1242/dev.116996
https://dev.biologists.org/content/142/5/910
から使わせていただきました。
図は本文中のものを使わせていただき、文章は私が本文を元に作製しました。
こだわったところ
見やすいように横A4にした
フォントは游明朝
編集しやすいようにWordで作成した
よって、まとめきれないときには複数ページにできる
PDF保存?紙保存?
このファイル、PDFで一つのフォルダに保存してもいいと思うんですが、ファイルを使うときは主に
新しいネタを考えるとき
やることなくてぼーっとしてるとき
くらいなんですよね。
そうじゃないときは、元論文に当たった方がたぶん速いです(笑)
なんで、いつでも手に取れるところに置いておき、パラパラと流し読みすることができることが大事だと思います。
まとめたものは、紙に印刷してファイルに閉じようと思います。

まとめ
書いてて、この記事つまんねーなとおもいました。
基本論文を見返すときは元論文に当たると思うので、まとめても見返さない気しかしません。
そもそも、論文って最小限に文章を圧縮して書いてあるものなので、それのSummaryなんて読みづらいものになるのは必然かな~。
読んだ論文数が10本くらいの学部生とかには結構効率的な勉強法になるかなとは思います。
これが余裕で書けるようになれば、30分/1論文くらいにスピードアップすることも夢じゃない(かも)!
HDDを1TBのSSDに換装した話
SSDっていいんですかね
SSD換装したのを記事にしようと思ってたのに後に後に伸ばしていたら、早半年、、、
せっかく写真とか取ったので、一応書きます。
PC初心者だったのですが、持ってたノートPCのハードディスクが壊れたのを機にSDD換装を行いました。
とりあえずつなぎとして知り合いから中古のHDDを格安で買い、サブPCからUSB回復diskを作成してWindows10をインストール。
データはすべてOnedriveに入れていたため助かりました。。。
バックアップ大事!!!京都大学の格安Office365最高!!!
またいつ壊れるかわからないので、大きめの容量の新品HDDを買おうかなと思っていたのですが、HDDと互換性のあるSSDというものが最近出てきていると聞き、調べてみると、、、
3倍以上違うらしいらしいですね。
SSDは
読み込み書き込みが速く
静音で
衝撃に強い
一方、
- 故障したときにデータが取り出せないことがある
らしいです。
僕は、データはすべてクラウド+HDDでバックアップをとっているので、完全にSSDに換装しても大丈夫かなと思いました。
CFD 1TB gamingとSumsung 860 EVO比較
AmazonでサーフィンしてSSD探してた時に出てきたのがこれ、「CFD 1TB CSSD-S6B01TMG4VT」。
今(2020/07/20現在)は生産停止しているらしいんですが、SSD 1TBで11,809円って結構破格だったんですね。
2019年に出た製品らしくレビューが(当時は)ほとんどなかったんですが、安さと好奇心で購入。
また、安全のためにレビューが多く、信用が置ける「Samsung 860 EVO 250GB」も購入して比較することにしました。
サブPCが160GBという激ヨワHDDだったので、2つのうち良い方をメインPC、ダメな方をサブPCに使おうという算段でした。
写真ありますよ
CFD 1TB CSSD-S6B01TMG4VT

初日に買ったもの(amazon)↑
 |
 |
|---|---|
| 表側 | 裏側 |
表側がかっこいい
Samsung 860 EVO 250GB
 |
 |
|---|---|
| ケース | 中身 |
 サブPCの方の分解写真(やってる感の演出)
サブPCの方の分解写真(やってる感の演出)
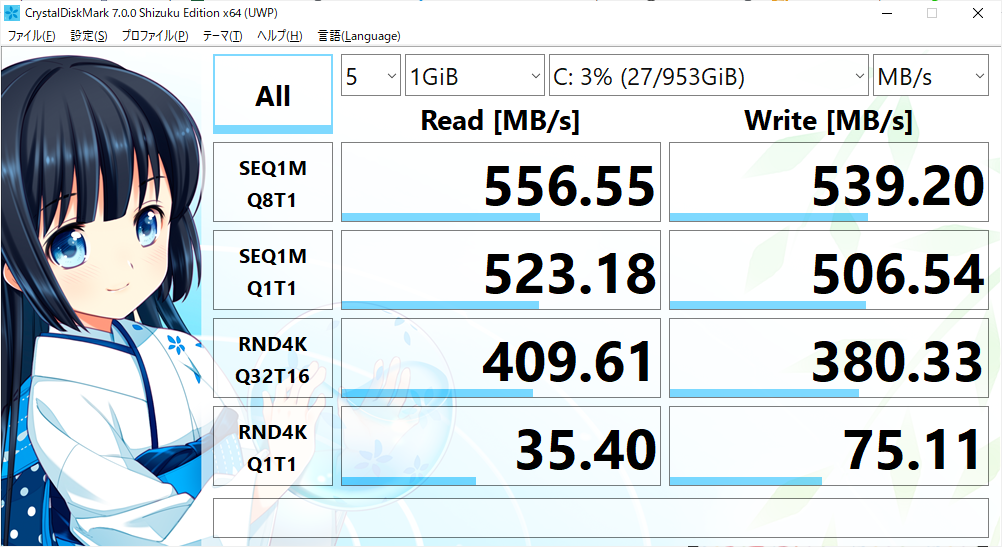
ずいぶん早くなりましたとさ(ベンチマークあり)
最近自分へのご褒美に買った、持ち運び用Surface go(128GB SSD) + 外付けSATA端子(↓)で速度を計測しました。
ソフトはcrystaldiskmark(sizuku edition)
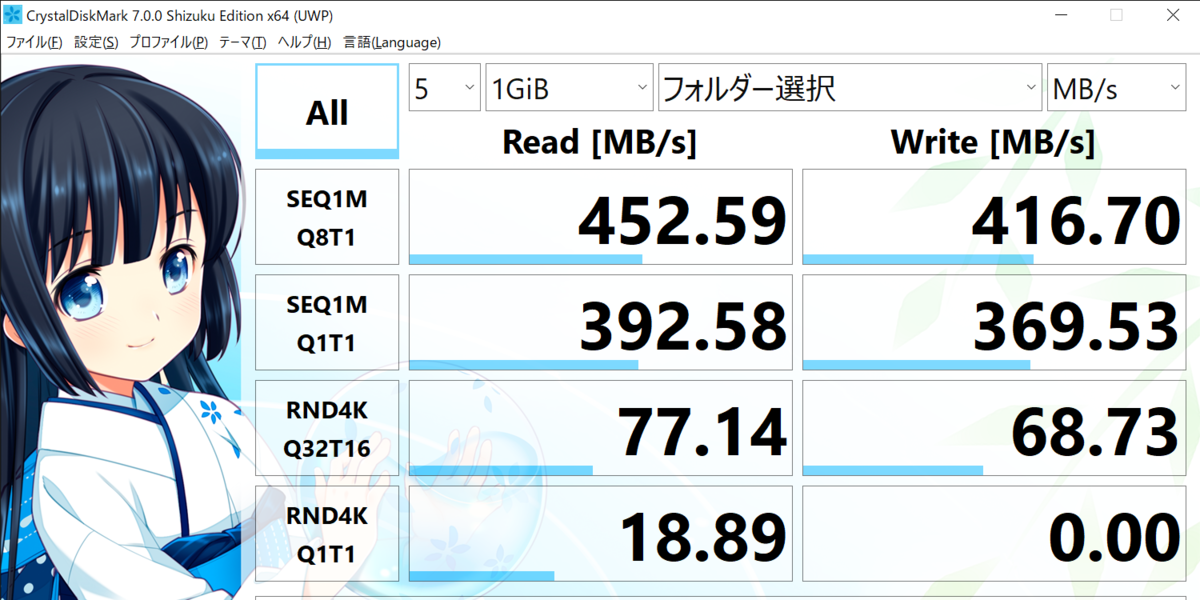
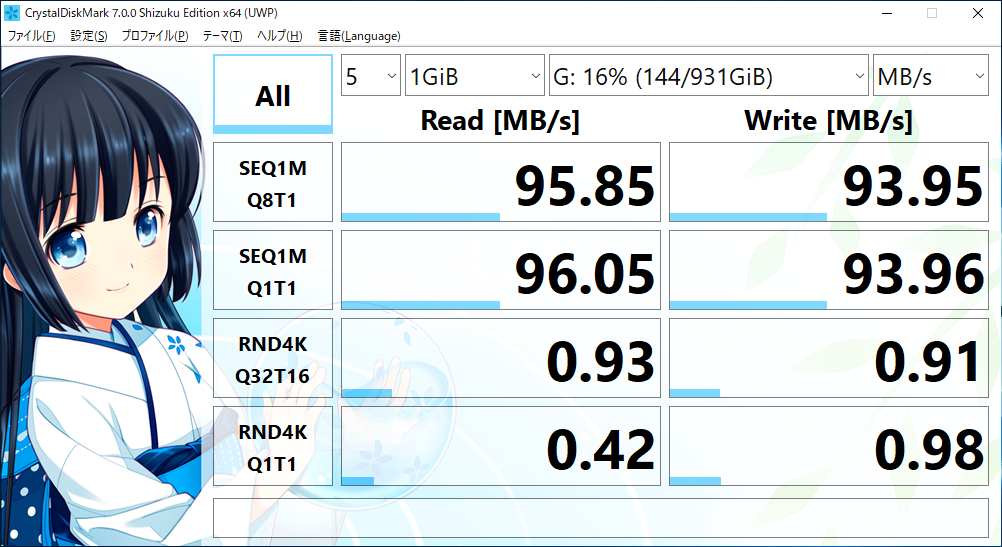
| CFD 1TB CSSD-S6B01TMG4VT | Samsung 860 EVO 250GB | WesternDigital 1TB HDD(2010年製) | Surface go内蔵SSD |
|---|---|---|---|
 |
 |
 |
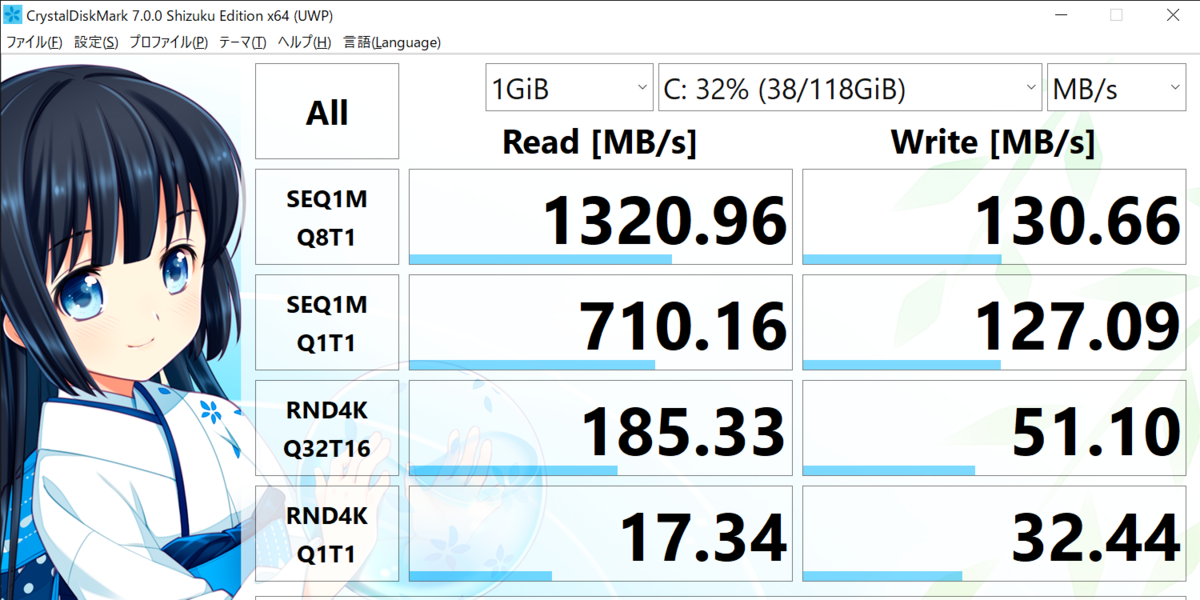 |
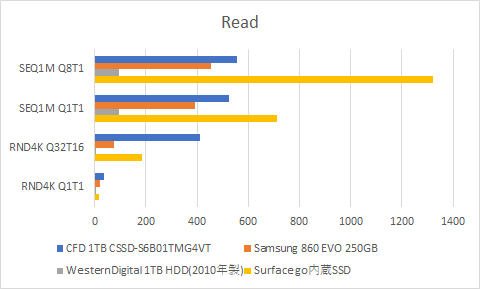
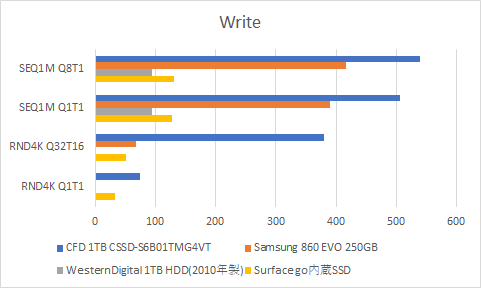
シーケンシャルRead&Writeでは5~6倍の差で、ランダムRead&Writeでは100倍を超える差が出るものもありました。
CFDとSamsungでは、全体的にCFDが良いというのもわかりました。やはり、gaming用のSSDは高い分性能がいいんですかね。ゲームしないんですけどね。
直近で買ったSurface go君があまり振るわなかったのに少しがっかりしました。シーケンシャルReadはめちゃくちゃ早いんですが、それ以外はすべてCFD SSDに負けてました。まあ、薄さと軽さで選んだので、別にいいですが。。。
まとめ
なんか、テーマが「SSD換装」なのか「ストレージ用ドライブ比較」なのかわかんなくなってしまいました。
参考になればうれしいです。
この記事書くときについでにMarkdown法の書き方とか、表の入れ方とか勉強できたので、次回以降もっと早くかけるといいな~
研究室のPCに画面共有する話~Anydesk編~
Anydeskってなんじゃらほい
以前、研究室のPCにTeamViewerで接続する方法について記事を書きました。
そのあと、twitterで知人から「うちはAnydesk使ってるで」と教えてもらい、ぼんやりと画面共有ソフトっていっぱいあるんやな~と思っておりました。
今日、うちの研究室のラボメンバーから「Anydeskってソフトめっちゃいいですよ」と勧められ、そんなにいいなら、、、と使ってみたら、めっちゃよかったという記事です。
Anydeskとは
AnyDeskは2014年にドイツで設立された、新しいリモートデスクトップアクセスのためのソフトウェア。これまでに、世界中で200 00万人以上のユーザーによりダウンロードされ、その後も毎月9 00万回のペースでダウンロードされ続けている。ソフトウェアAnyDeskは、当社開発のコーデックDeskRTに基づき、オフィスからでも世界の反対側からでも、実質的にタイムラグのないオンラインでの共同作業を可能にした。ドイツで最も成長が著しい50社のうちの1社に選ばれる。
らしいです。
引用元↓
TeamViewerの後続らしく、ほぼ同じような機能を持っているといわれています。
比較している人↓
これ、、、TeamViewerの上位互換???
いんすとーる
インストール方法は、以下のURLのAnydeskのサイトで「無料ダウンロード」ボタンを押すだけでexeファイルがダウンロードされます。
このexeファイル、すでにソフトとして機能するので、「他のワークスペース」の欄に接続先のワークスペースを入れればすぐに接続できます。
また、PC内にソフトをインストールすることもできます。 exeファイルとあまりできることは変わらないのですが、ソフトの自動更新をしてくれたり、画面共有越しに印刷できるようになったりするようです。
Program Filesを汚したくない人などは、インストールなしで使ってもいいかもしれません。
PCにインストールしたい場合は「Anydeskをインストールする」をクリックしてダイアログに従っていけばインストールできます。

パスワード設定
また、そのままではクライアント側からリモート側に接続する際にリモート側で許可処理を行わないといけないのですが、パスワードを設定すればリモート側での処理をしなくても画面共有できるようになります。

パスワードを変更する→セキュリティ→不在アクセス→無人アクセスを許可する
のチェックを付けて、「ワークスペースへのパスワードを変更する...」からパスワードを設定しましょう。
知らないデバイスからの検索除外
もう一つのおすすめは
マスワードを変更する→セキュリティ
の一番下にある「検索結果」の欄の
・ローカルネットワーク内のほかのAnydeskクライアントを検索する
のチェックを外し、
・検索結果からこのデバイスを除外する
のチェックを付けることです。

これをすることで、他のPCから間違って接続申請が来たりすることはなくなります。
また、一度つないだことのあるクライアントの接続は思えておいてくれるので、最初だけIDを直打ちして接続しておけば、特に不便はありません。
メリット
羅列していきます。
画面共有先でAlt+Tabなどのショートカットキーができる(Control+CとかはTeamViewerでもできるが、Anydeskの方が圧倒的に多い)
速さ(普通にめっちゃ早い。TeamViewerと同じレベル)
画面共有中に端っこにソフトが映ったりしない
UIがわかりやすい(ファイル共有など、使いそうな機能のボタンが上部にまとまっている)
商用ライセンスガーとかいわれない!!!
デメリット
- VPNでローカルにアクセス→ローカルIP指定での接続というのができるのかできないのかの情報がなかった。
一応、アドレス入力欄にローカルIPを入れるとつながるのですが、これが本当にセキュアな状態なのかどうかの情報がありませんでした。
人によってはセキュアじゃないからちょっと、、、とかいうこともあるかもしれません。
これからはAnydeskにします
僕は、ファイル自動公衆送信機能を持たなければP2P使ってもいい大学にいるので、これからはAnydeskにします。
実験マウスを管理するための専用フォームを開発する5-最終回-
概要
以前から「マウス管理システム」なるものをちょくちょく作っています。
第一回
第二回
第三回
第四回
これを、さらに変更しました。
変更点としては、
gsファイルとCSSファイルを変更して、スマホでちょうどよく表示できるようにした(gasアプリの特性として、metaタグをいじるだけではスマホ画面に合わせることができなかったため、doGet()をいじりました。)
屠殺用ページのマウス選択を、selectタブで選ぶ形式から表のボタンを押す形式に変えました。(ユーザーの体感が早くなると同時に、屠殺する匹数を選べるようになりました。)
屠殺したマウスを「屠殺マウス」シートに記録できるようにしました。
仔分け用のページを作成しました。
交配用のページを作成しました。
resultのページを削除し、入力が終わったら手動でトップページに戻るようにした(たまにgoogle.script.runコマンドが回りきる前にページ遷移してしまって、うまく入力が反映されないことがあったからです)。
を主にいじりました。
できたもの
完成版アプリはこちら↓(僕の本番用は別ですので、自由に使っていただいて構いません)
スプレッドシートは引き続き以下のものを使用しています↓(これも練習用ですのでいじってもらって構いません)
コードはgithubにまとめてバージョン管理するようにしました。
使用方法
まず、マウスが生まれたときです。 トップページで「生まれた」をクリックすれば、生まれたマウスを入力するページに遷移します。 生まれたオス、メスの匹数をプルダウンで入力し、「今日」から「7日前」までの間で生まれた日を指定し、出産した母マウスを選んだ後に「送信する」ボタンを押します(映像にはプルダウンが表示されていませんが、録画の使用なので実際は表示されます)。 入力が終わると、ポップアップが出ます。
次に、マウスを実験などで屠殺した時です。 トップページで「屠殺した」をクリックし、「使用したマウス」のプルタブで使用したマウスが記入されているスプレッドシートのシート名を入れます。 その後、使用した数だけ「追加」ボタンを押し、使用した理由を下の入力欄に記入した後、「送信する」ボタンを押します。
https://youtu.be/zwJqOiqgP3Myoutu.be
次に、マウスが3週齢になった時のオスメスの仔分けです(他の研究室がなんと呼んでるかわかりませんが笑)。 トップページで「仔分け」をクリックし、「仔分けしたマウス」のリストの中から仔分けしたマウスを選び、チェックを入れます。 下の「匹数の変更」の欄で、生まれた時と仔分け時に数が異なってしまったものを調節し、「間引き」の欄で数が多すぎて間引くマウスを入力します。 すべて入力したら「送信する」ボタンを押します。
最後に、マウス同士で交配をかける操作です。 トップページで「交配」をクリックし、最初のプルタブで「交配開始」か「交配終了」を選びます。 下のリストでは、交配をかけることのできるオス、メスが表示されていますので、交配をかけるもの同士にチェックを入れ、「送信する」ボタンを押します。
あとがき
やっと使用に耐えうるものができました。
最後できたと思ったら、入力が反映される時と反映されない時があってビビりました笑
結局、google.script.runコマンドの速度とページ遷移の速度の兼ね合いでしたね。
GAS難しい。
2020/12/03追記
記事をまとめました。